Mplusでの平行分析の実行の仕方は以前に書いたが、Rでも実行できる。
今回もbfiデータの1~25列目を使用する(参考: http://ides.hatenablog.com/entry/2019/03/19/093726)。
library("psych")
data(bfi)
d1<-bfi[1:25]
平行分析は次のように指定する。
fa.parallel(d1)
"Parallel analysis suggests that the number of factors = 6 and the number of components = 6"と出力され6因子、6成分と提案があった。 今まで行ってきた他の方法よりも因子数が多い印象である。
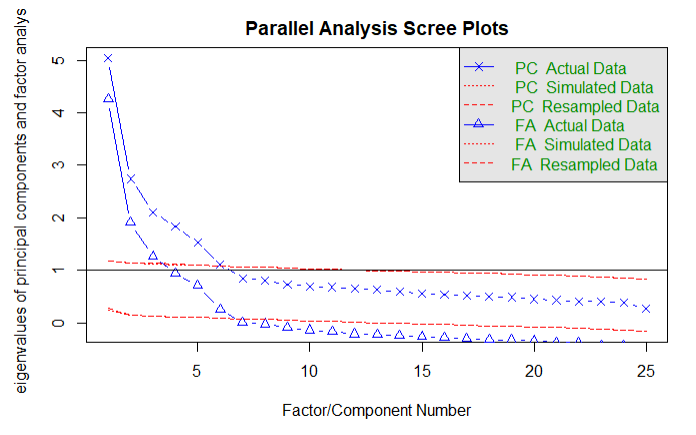
ちなみに図にあるPCとは" principal components"のことで、主成分分析FAはprincipal axis factor analysisで主因子法のことである。
オプションとしてfa="minres"のように表記して、minres(最小残差法), ml(最尤法), uls(重みづけのない最小二乗法), wls(重み付き最小2乗法), gls(一般化最小二乗法), pa(主成分)が指定できる。デフォルトはminresである。
対角SMC平行分析はオプションで指定を行う。
fa.parallel(d1, SMC=TRUE)
こちらは8因子を提案がされてきた。
堀啓造先生によると下記のような特徴があるらしい。
対角に SMC 入れた分析(SMC-EIGEN)の場合、ほかの推定法に比べ極端に多くの因子数を推測する。
対角SMC の平行分析はその点それ以上の因子をとってはいけないという指標となりうるのである。
欠点としてはマイナー因子も拾うことがあるので,まったく因子を想定しない項目群では多めの因子数を推定する。
https://www.ec.kagawa-u.ac.jp/~hori/yomimono/nfac.ppt
たしかに今回のシミュレーションでも最も多い因子数を提案してきた。