lavaanで測定普遍性を確認する方法。
rstudio-pubs-static.s3.amazonaws.com
ある構成要素についてグループ間で比較を行うとき、暗黙のうちに測定の不変性を仮定している。回帰分析、t検定、混合効果モデルなどを行っている時も、その構成要素が同じように機能するであろうという仮定の下での分析になっている。この仮定を確かなものにするための一つの方法が、測定不変性を確認することである。ただし、間接的な方法なので、根本的なアプローチかというとそうでもないところには注意が必要であるが、測定普遍性を確認し、グループ間の違いについて論じる際には、言及が必要となる。
データ
古典的なHolzinger and Swineford (1939)のデータセット。2つの異なる学校(PasteurとGrant-White)の7年生と8年生の子供たちの知能テストのスコアである。オリジナルのデータセット(MBESSパッケージで利用可能)では、26のテストのスコアがある。lavaanで利用できるのは、9つの変数を持つより小さなサブセットであり、このデータセットがより広く使われている(たとえば、Joreskogの1969年の論文では、Grant-Whiteの学校からの145人の被験者のみを使用している)。
library("lavaan")
data("HolzingerSwineford1939")
head(HolzingerSwineford1939)
id sex ageyr agemo school grade x1 x2 x3 x4 x5 x6 x7 x8 x9 1 1 1 13 1 Pasteur 7 3.333333 7.75 0.375 2.333333 5.75 1.2857143 3.391304 5.75 6.361111 2 2 2 13 7 Pasteur 7 5.333333 5.25 2.125 1.666667 3.00 1.2857143 3.782609 6.25 7.916667 3 3 2 13 1 Pasteur 7 4.500000 5.25 1.875 1.000000 1.75 0.4285714 3.260870 3.90 4.416667 4 4 1 13 2 Pasteur 7 5.333333 7.75 3.000 2.666667 4.50 2.4285714 3.000000 5.30 4.861111 5 5 2 12 2 Pasteur 7 4.833333 4.75 0.875 2.666667 4.00 2.5714286 3.695652 6.30 5.916667 6 6 2 14 1 Pasteur 7 5.333333 5.00 2.250 1.000000 3.00 0.8571429 4.347826 6.65 7.500000
x1, x2, x3 は視覚的要因、x4, x5, x6 はテキスト要因、x7, x8, x9はスピード要因である。
確証的因子分析(CFA)
最もシンプルなCFAは以下のものである。
HS.model <- ' visual =~ x1 + x2 + x3
textual =~ x4 + x5 + x6
speed =~ x7 + x8 + x9 '
fit <- cfa(HS.model, data=HolzingerSwineford1939)
summary(fit, fit.measures=TRUE)
fitMeasures(fit, c("chisq","df","pvalue","gfi","agfi","cfi","tli","rmsea", "srmr"))
フィッシング指標のみ示しておこう。
chisq df pvalue gfi agfi cfi tli rmsea srmr 85.306 24.000 0.000 0.943 0.894 0.931 0.896 0.092 0.065
悪くはないがいまいちなところもある。
構成モデル Configural model
最初のステップ。モデルが、グループのそれぞれに適合することを示すことである。ここでのグループは学校ごとである。group = "school"を追加する。
configural <- cfa(HS.model, data=HolzingerSwineford1939, group = "school")
summary(configural, fit.measures=TRUE)
fitMeasures(configural, c("chisq","df","pvalue","gfi","agfi","cfi","tli","rmsea", "srmr"))
こちらもフィッシング指標のみ示しておこう。
chisq df pvalue gfi agfi cfi tli rmsea srmr 115.851 48.000 0.000 0.995 0.989 0.923 0.885 0.097 0.068
やはりいまいちである。
弱不変性 Weak Invariance
測定基準不変性 metric invarianceのことである。
因子負荷がグループ間で等しくなるように制約するモデルである。これは因子がグループ間で同じ有意性を持っていることを仮定している。
weak.invariance <- cfa(HS.model, data=HolzingerSwineford1939, group = "school", group.equal = "loadings")
summary(weak.invariance, fit.measures = TRUE)
fitMeasures(weak.invariance, c("chisq","df","pvalue","gfi","agfi","cfi","tli","rmsea", "srmr"))
こちらもフィッシング指標のみ示しておこう。
chisq df pvalue gfi agfi cfi tli rmsea srmr 124.044 54.000 0.000 0.995 0.989 0.921 0.895 0.093 0.072
尤度比検定 構成モデルvs.弱不変性
lavTestLRT(weak.invariance, configural)
少し前まではanova(a,b)で動作していたようだが、現在はlavTestLRTに変更されている。
Chi-Squared Difference Test
Df AIC BIC Chisq Chisq diff Df diff Pr(>Chisq)
configural 48 7484.4 7706.8 115.85
weak.invariance 54 7480.6 7680.8 124.04 8.1922 6 0.2244
帰無仮説は、構成モデルは弱い不変性モデルよりも適合性が向上していないというものだ。P=0.2244であったため有意だとは認められず、帰無仮説は棄却されない。これは、弱い不変性があることの証拠である。測定普遍性を示す場合、P<.05になるとマズいということだ。
フィッシング指標をまとめるコードもあったので掲載しておこう。
fit.stats <- rbind(fitmeasures(configural, fit.measures = c("chisq", "df", "rmsea", "tli", "cfi", "aic")),
fitmeasures(weak.invariance, fit.measures = c("chisq", "df", "rmsea", "tli", "cfi", "aic")))
rownames(fit.stats) <- c("configural", "weak invariance")
fit.stats
内容は必要なものを入れて使うとよいだろう。
chisq df rmsea tli cfi aic configural 115.8513 48 0.09691486 0.8850976 0.9233984 7484.395 weak invariance 124.0435 54 0.09283654 0.8945646 0.9209235 7480.587
強不変性 strong invariance
尺度妥当性scalar invarianceのことである。
強不変性は切片がグループ間で等しいという制約を追加する。これは、構成要素(因子負荷)の有意味性と、基礎となる顕在変数(切片)のレベルが、両方のグループで等しいというモデルである。具体的にはgroup.equal = c( "loadings", "intercepts")を追加する。loadingsの代わりにinterceptsを追加するのでは、両方とも入れる。
strong.invariance <- cfa(HS.model, data=HolzingerSwineford1939, group = "school",
group.equal = c( "loadings", "intercepts"))
summary(strong.invariance, fit.measures = TRUE)
fitMeasures(strong.invariance, c("chisq","df","pvalue","gfi","agfi","cfi","tli","rmsea", "srmr"))
こちらもフィッシング指標のみ示しておこう。
chisq df pvalue gfi agfi cfi tli rmsea srmr 164.103 60.000 0.000 0.993 0.987 0.882 0.859 0.107 0.082
視覚、テキスト、スピードの潜在平均が推定されている。強不変性があるかどうかをテストするまでは、グループ間で比較すべきではないとされている。
尤度比検定 弱不変性vs. 強不変性
lavaanはベータ版と宣言しているようにコマンドが時々変わり、尤度比検定のコマンドが変更されている(参照)
lavTestLRT(strong.invariance, weak.invariance)
Chi-Squared Difference Test
Df AIC BIC Chisq Chisq diff Df diff Pr(>Chisq)
weak.invariance 54 7480.6 7680.8 124.04
strong.invariance 60 7508.6 7686.6 164.10 40.059 6 4.435e-07 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
P<.05であるため、帰無仮説が棄却される。
弱不変モデルが強不変モデルよりもよく適合しているようだ。弱不変モデルの方がAICとBICが低いことからも明らかである。
次にすることは部分不変性partial invarianceがあるかどうかを調べることである。lavaanのlavTestScore()を使用することで確認できる。この関数を使うことで、グループ間の等値制約を解放するべき場所を見つけることができる。modindices()は、新しいパスに関連して新たに追加されたパラメータの修正インデックスのみを表示する。
lavTestScore(strong.invariance)
$test
total score test:
test X2 df p.value
1 score 46.956 15 0
$uni
univariate score tests:
lhs op rhs X2 df p.value
1 .p2. == .p38. 0.306 1 0.580
2 .p3. == .p39. 1.636 1 0.201
3 .p5. == .p41. 2.744 1 0.098
4 .p6. == .p42. 2.627 1 0.105
5 .p8. == .p44. 0.027 1 0.871
6 .p9. == .p45. 0.004 1 0.952
7 .p25. == .p61. 5.847 1 0.016
8 .p26. == .p62. 6.863 1 0.009
9 .p27. == .p63. 19.193 1 0.000
10 .p28. == .p64. 2.139 1 0.144
11 .p29. == .p65. 1.563 1 0.211
12 .p30. == .p66. 0.032 1 0.857
13 .p31. == .p67. 15.021 1 0.000
14 .p32. == .p68. 4.710 1 0.030
15 .p33. == .p69. 1.498 1 0.221
pはparTable()を使うと具体的に確認できる。
options(max.print=100000) parTable(strong.invariance)
id lhs op rhs user block group free ustart exo label plabel start est se 1 1 visual =~ x1 1 1 1 0 1 0 .p1. 1.000 1.000 0.000 2 2 visual =~ x2 1 1 1 1 NA 0 .p2. .p2. 0.769 0.576 0.101 3 3 visual =~ x3 1 1 1 2 NA 0 .p3. .p3. 1.186 0.798 0.112 4 4 textual =~ x4 1 1 1 0 1 0 .p4. 1.000 1.000 0.000 5 5 textual =~ x5 1 1 1 3 NA 0 .p5. .p5. 1.237 1.120 0.066 6 6 textual =~ x6 1 1 1 4 NA 0 .p6. .p6. 0.865 0.932 0.056 7 7 speed =~ x7 1 1 1 0 1 0 .p7. 1.000 1.000 0.000 8 8 speed =~ x8 1 1 1 5 NA 0 .p8. .p8. 1.227 1.130 0.145 9 9 speed =~ x9 1 1 1 6 NA 0 .p9. .p9. 0.827 1.009 0.132 10 10 x1 ~~ x1 0 1 1 7 NA 0 .p10. 0.698 0.555 0.139 11 11 x2 ~~ x2 0 1 1 8 NA 0 .p11. 0.752 1.296 0.158 12 12 x3 ~~ x3 0 1 1 9 NA 0 .p12. 0.673 0.944 0.136 13 13 x4 ~~ x4 0 1 1 10 NA 0 .p13. 0.660 0.445 0.069 14 14 x5 ~~ x5 0 1 1 11 NA 0 .p14. 0.854 0.502 0.082 15 15 x6 ~~ x6 0 1 1 12 NA 0 .p15. 0.487 0.263 0.050 16 16 x7 ~~ x7 0 1 1 13 NA 0 .p16. 0.585 0.888 0.120 17 17 x8 ~~ x8 0 1 1 14 NA 0 .p17. 0.476 0.541 0.095 18 18 x9 ~~ x9 0 1 1 15 NA 0 .p18. 0.489 0.654 0.096 19 19 visual ~~ visual 0 1 1 16 NA 0 .p19. 0.050 0.796 0.172 20 20 textual ~~ textual 0 1 1 17 NA 0 .p20. 0.050 0.879 0.131 21 21 speed ~~ speed 0 1 1 18 NA 0 .p21. 0.050 0.322 0.082 22 22 visual ~~ textual 0 1 1 19 NA 0 .p22. 0.000 0.410 0.095 23 23 visual ~~ speed 0 1 1 20 NA 0 .p23. 0.000 0.178 0.066 24 24 textual ~~ speed 0 1 1 21 NA 0 .p24. 0.000 0.180 0.062 25 25 x1 ~1 0 1 1 22 NA 0 .p25. .p25. 4.941 5.001 0.090 26 26 x2 ~1 0 1 1 23 NA 0 .p26. .p26. 5.984 6.151 0.077 27 27 x3 ~1 0 1 1 24 NA 0 .p27. .p27. 2.487 2.271 0.083 28 28 x4 ~1 0 1 1 25 NA 0 .p28. .p28. 2.823 2.778 0.087 29 29 x5 ~1 0 1 1 26 NA 0 .p29. .p29. 3.995 4.035 0.096 30 30 x6 ~1 0 1 1 27 NA 0 .p30. .p30. 1.922 1.926 0.079 31 31 x7 ~1 0 1 1 28 NA 0 .p31. .p31. 4.432 4.242 0.073 32 32 x8 ~1 0 1 1 29 NA 0 .p32. .p32. 5.563 5.630 0.072 33 33 x9 ~1 0 1 1 30 NA 0 .p33. .p33. 5.418 5.465 0.069 34 34 visual ~1 0 1 1 0 0 0 .p34. 0.000 0.000 0.000 35 35 textual ~1 0 1 1 0 0 0 .p35. 0.000 0.000 0.000 36 36 speed ~1 0 1 1 0 0 0 .p36. 0.000 0.000 0.000 37 37 visual =~ x1 1 2 2 0 1 0 .p37. 1.000 1.000 0.000 38 38 visual =~ x2 1 2 2 31 NA 0 .p2. .p38. 0.896 0.576 0.101 39 39 visual =~ x3 1 2 2 32 NA 0 .p3. .p39. 1.155 0.798 0.112 40 40 textual =~ x4 1 2 2 0 1 0 .p40. 1.000 1.000 0.000 41 41 textual =~ x5 1 2 2 33 NA 0 .p5. .p41. 0.991 1.120 0.066 42 42 textual =~ x6 1 2 2 34 NA 0 .p6. .p42. 0.962 0.932 0.056 43 43 speed =~ x7 1 2 2 0 1 0 .p43. 1.000 1.000 0.000 44 44 speed =~ x8 1 2 2 35 NA 0 .p8. .p44. 1.282 1.130 0.145 45 45 speed =~ x9 1 2 2 36 NA 0 .p9. .p45. 0.895 1.009 0.132 46 46 x1 ~~ x1 0 2 2 37 NA 0 .p46. 0.659 0.654 0.128 47 47 x2 ~~ x2 0 2 2 38 NA 0 .p47. 0.613 0.964 0.123 48 48 x3 ~~ x3 0 2 2 39 NA 0 .p48. 0.537 0.641 0.101 49 49 x4 ~~ x4 0 2 2 40 NA 0 .p49. 0.629 0.343 0.062 50 50 x5 ~~ x5 0 2 2 41 NA 0 .p50. 0.671 0.376 0.073 51 51 x6 ~~ x6 0 2 2 42 NA 0 .p51. 0.640 0.437 0.067 52 52 x7 ~~ x7 0 2 2 43 NA 0 .p52. 0.531 0.625 0.095 53 53 x8 ~~ x8 0 2 2 44 NA 0 .p53. 0.547 0.434 0.088 54 54 x9 ~~ x9 0 2 2 45 NA 0 .p54. 0.526 0.522 0.086 55 55 visual ~~ visual 0 2 2 46 NA 0 .p55. 0.050 0.708 0.160 56 56 textual ~~ textual 0 2 2 47 NA 0 .p56. 0.050 0.870 0.131 57 57 speed ~~ speed 0 2 2 48 NA 0 .p57. 0.050 0.505 0.115 58 58 visual ~~ textual 0 2 2 49 NA 0 .p58. 0.000 0.427 0.097 59 59 visual ~~ speed 0 2 2 50 NA 0 .p59. 0.000 0.329 0.082 60 60 textual ~~ speed 0 2 2 51 NA 0 .p60. 0.000 0.236 0.073 61 61 x1 ~1 0 2 2 52 NA 0 .p25. .p61. 4.930 5.001 0.090 62 62 x2 ~1 0 2 2 53 NA 0 .p26. .p62. 6.200 6.151 0.077 63 63 x3 ~1 0 2 2 54 NA 0 .p27. .p63. 1.996 2.271 0.083 64 64 x4 ~1 0 2 2 55 NA 0 .p28. .p64. 3.317 2.778 0.087 65 65 x5 ~1 0 2 2 56 NA 0 .p29. .p65. 4.712 4.035 0.096 66 66 x6 ~1 0 2 2 57 NA 0 .p30. .p66. 2.469 1.926 0.079 67 67 x7 ~1 0 2 2 58 NA 0 .p31. .p67. 3.921 4.242 0.073 68 68 x8 ~1 0 2 2 59 NA 0 .p32. .p68. 5.488 5.630 0.072 69 69 x9 ~1 0 2 2 60 NA 0 .p33. .p69. 5.327 5.465 0.069 70 70 visual ~1 0 2 2 61 NA 0 .p70. 0.000 -0.148 0.122 71 71 textual ~1 0 2 2 62 NA 0 .p71. 0.000 0.576 0.117 72 72 speed ~1 0 2 2 63 NA 0 .p72. 0.000 -0.177 0.090 73 73 .p2. == .p38. 2 0 0 0 NA 0 0.000 0.000 0.000 74 74 .p3. == .p39. 2 0 0 0 NA 0 0.000 0.000 0.000 75 75 .p5. == .p41. 2 0 0 0 NA 0 0.000 0.000 0.000 76 76 .p6. == .p42. 2 0 0 0 NA 0 0.000 0.000 0.000 77 77 .p8. == .p44. 2 0 0 0 NA 0 0.000 0.000 0.000 78 78 .p9. == .p45. 2 0 0 0 NA 0 0.000 0.000 0.000 79 79 .p25. == .p61. 2 0 0 0 NA 0 0.000 0.000 0.000 80 80 .p26. == .p62. 2 0 0 0 NA 0 0.000 0.000 0.000 81 81 .p27. == .p63. 2 0 0 0 NA 0 0.000 0.000 0.000 82 82 .p28. == .p64. 2 0 0 0 NA 0 0.000 0.000 0.000 83 83 .p29. == .p65. 2 0 0 0 NA 0 0.000 0.000 0.000 84 84 .p30. == .p66. 2 0 0 0 NA 0 0.000 0.000 0.000 85 85 .p31. == .p67. 2 0 0 0 NA 0 0.000 0.000 0.000 86 86 .p32. == .p68. 2 0 0 0 NA 0 0.000 0.000 0.000 87 87 .p33. == .p69. 2 0 0 0 NA 0 0.000 0.000 0.000
total score test:として出力されているのはラグランジュ乗数検定である。これは、等値制約を解放することによって、ベース・モデルに対する適合性の改善を表すかどうかの検定である。カイ2乗差検定で帰無仮説が棄却されるものを探す。カイ2乗差(X2)で最大の変化を持つのはx3(.p27 == .p63)である(カイ二乗値19.193であり、P=0.000である)。ここの切片を解放すべきであることがわかる。最初にこの点をモデルへ反映させよう。
部分的強不変性を導くステップ1
先の切片の等値制約を外すのはx3~x1であるため、group.partial = c("x3 ~ 1")を指定する。
strong.invariance.x3 <- cfa(HS.model, data=HolzingerSwineford1939, group = "school",
group.equal = c( "loadings", "intercepts"),
group.partial = c("x3 ~ 1"))
lavTestScore(strong.invariance.x3)
結果。
$test
total score test:
test X2 df p.value
1 score 27.528 14 0.016
$uni
univariate score tests:
lhs op rhs X2 df p.value
1 .p2. == .p38. 0.734 1 0.392
2 .p3. == .p39. 0.485 1 0.486
3 .p5. == .p41. 2.760 1 0.097
4 .p6. == .p42. 2.630 1 0.105
5 .p8. == .p44. 0.026 1 0.872
6 .p9. == .p45. 0.002 1 0.960
7 .p25. == .p61. 2.833 1 0.092
8 .p26. == .p62. 2.833 1 0.092
9 .p28. == .p64. 2.136 1 0.144
10 .p29. == .p65. 1.560 1 0.212
11 .p30. == .p66. 0.032 1 0.857
12 .p31. == .p67. 15.023 1 0.000
13 .p32. == .p68. 4.727 1 0.030
14 .p33. == .p69. 1.492 1 0.222
ラグランジュ検定が棄却されているので、さらに等値制約の解放が必要であることがわかる。
次に最大のカイ2乗差(X2)を持つのはx7 (.p31. == .p67.)であるため、モデルをさらに改変する。
ステップ2
strong.invariance.x3x7 <- cfa(HS.model, data=HolzingerSwineford1939, group = "school",
group.equal = c( "loadings", "intercepts"),
group.partial = c("x3 ~ 1", "x7 ~ 1"))
lavTestScore(strong.invariance.x3x7)
$test
total score test:
test X2 df p.value
1 score 12.583 13 0.481
$uni
univariate score tests:
lhs op rhs X2 df p.value
1 .p2. == .p38. 0.734 1 0.391
2 .p3. == .p39. 0.492 1 0.483
3 .p5. == .p41. 2.769 1 0.096
4 .p6. == .p42. 2.631 1 0.105
5 .p8. == .p44. 0.013 1 0.910
6 .p9. == .p45. 0.062 1 0.803
7 .p25. == .p61. 2.832 1 0.092
8 .p26. == .p62. 2.832 1 0.092
9 .p28. == .p64. 2.135 1 0.144
10 .p29. == .p65. 1.563 1 0.211
11 .p30. == .p66. 0.032 1 0.858
12 .p32. == .p68. 0.053 1 0.818
13 .p33. == .p69. 0.053 1 0.818
P=0.481となったため、ラグランジュ検定が棄却されていないことを確認。
次に行うのは、弱不変性モデルとの比較である。切片に等値制約を掛けた場合、P<.05になっていたので、部分的強不変モデルを作成したわけだが、この作成したモデルが弱不変性モデルと比べて改善できているかを確認する。
lavTestLRT(strong.invariance.x3x7, weak.invariance)
結果。
Chi-Squared Difference Test
Df AIC BIC Chisq Chisq diff Df diff Pr(>Chisq)
weak.invariance 54 7480.6 7680.8 124.04
strong.invariance.x3x7 58 7478.0 7663.3 129.42 5.3789 4 0.2506
P<.05ではないため、測定不変性が確認できている。ちなみに、このコマンドはanova(a,b)がまだ通るようである(不思議)。
fit.stats2 <- rbind(fitmeasures(strong.invariance, fit.measures = c("chisq", "df", "rmsea", "tli", "cfi", "aic")),
fitmeasures(strong.invariance.x3x7, fit.measures = c("chisq", "df", "rmsea", "tli", "cfi", "aic")))
rownames(fit.stats2) <- c("strong", "strong with x3 x7")
fit.stats <- rbind(fit.stats, fit.stats2)
round(fit.stats, 4)
フィッティング指標をモデルごとに比較してみよう。
chisq df rmsea tli cfi aic configural 115.8513 48 0.0969 0.8851 0.9234 7484.395 weak invariance 124.0435 54 0.0928 0.8946 0.9209 7480.587 strong 164.1028 60 0.1074 0.8590 0.8825 7508.646 strong with x3 x7 129.4225 58 0.0905 0.8999 0.9194 7477.966
summary(strong.invariance.x3x7)
切片の一部だけ表示。
Group 1 [Pasteur]:
Intercepts:
Estimate Std.Err Z-value P(>|z|)
visual 0.000
textual 0.000
speed 0.000
Group 2 [Grant-White]:
Intercepts:
Estimate Std.Err Z-value P(>|z|)
visual 0.051 0.129 0.393 0.695
textual 0.576 0.117 4.918 0.000
speed -0.071 0.089 -0.800 0.424
テキストについては強不変性があり、視覚と速度の両方の要因については部分的に強不変性があることがわかった。学校2(Grant-White)の生徒が、学校1(Pasteur)よりもテキストの潜在的構成要素で優れていることが判明した。彼らはPasteurの生徒よりも0.576高くなると予想されます。部分的な強不変性を許容すると、Grant-Whiteの生徒とPasteurの生徒の間には、視覚と速度に関して差がない。
厳密不変性 Strict Invariance
厳密不変性はグループ間の顕在変数の残差分散が等しいという制約を追加する。今までの分析で部分的不変性しか確認できなかったため、あるべきパラメータ(たとえば、x3 と x7 の切片)を自由にして、残差分散と同様に、他のすべてのパラメータ(すなわち、因子負荷量と切片)を制限することになる。raw score に基づく比較を行いたい場合(例えば,変数のスコアの合計に基づく)我々は厳密不変性を必要になる。 これは,観察された分散が真のスコア分散と残差/誤差分散の合計であるからである。残差分散が同じであれば、それらは同量の真のスコア分散を持ちます。前と同様に潜在平均を推定する。
等値制約に"residuals"を追加する。
strict.invariance.x3x7 <- cfa(HS.model, data=HolzingerSwineford1939, group = "school",
group.equal = c( "loadings", "intercepts", "residuals"),
group.partial = c("x3 ~ 1", "x7 ~ 1"))
anova(strong.invariance.x3x7, strict.invariance.x3x7)
こちらはlavTestLRT(a,b)は通らず、anova(a,b)しか通らないので注意。そのうち通るようになるのだろうが。
Chi-Squared Difference Test
Df AIC BIC Chisq Chisq diff Df diff Pr(>Chisq)
strong.invariance.x3x7 58 7478.0 7663.3 129.42
strict.invariance.x3x7 67 7477.8 7629.8 147.26 17.838 9 0.0371 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
カイ二乗差検定は有意だが、P値が.037というのは、それほど違和感があるわけではない。他の適合尺度を見よう。
fit.stats <- rbind(fit.stats, fitmeasures(strict.invariance.x3x7, fit.measures = c("chisq", "df", "rmsea", "tli", "cfi", "aic")))
rownames(fit.stats)[5] <- "strict invariance"
round(fit.stats, 4)
chisq df rmsea tli cfi aic configural 115.8513 48 0.0969 0.8851 0.9234 7484.395 weak invariance 124.0435 54 0.0928 0.8946 0.9209 7480.587 strong 164.1028 60 0.1074 0.8590 0.8825 7508.646 strong with x3 x7 129.4225 58 0.0905 0.8999 0.9194 7477.966 strict invariance 147.2605 67 0.0892 0.9026 0.9094 7477.804
厳密不変性は、x3 と x7 の切片が自由である強い不変性よりも、限定的な改善であることがわかる。完全な厳密不変性があれば、顕在変数がグループ間で等しく信頼できると言えるだろう。これは、平均の比較やグループ間の回帰を実行するために、合計スコアを使用できることを意味する。完全な厳密不変性を持っていないので、この性質で比較できる唯一の尺度は、テキスト要因である。構造モデルについて述べるときは、この問題に戻り、構造的不変性、具体的には因子分散、因子共分散、SEMにおける回帰係数について議論する。
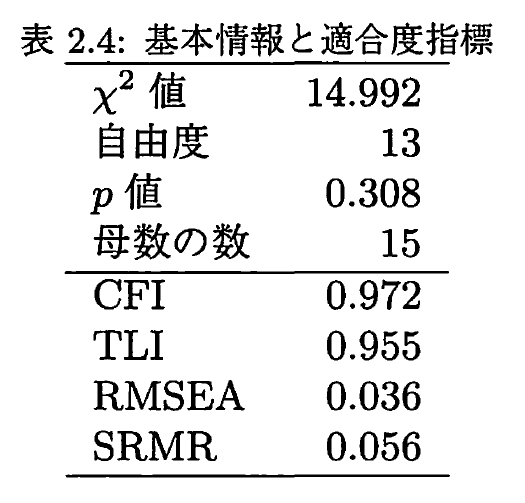
実用例
こちらの文献では次のような表記がされている。
Adaptation of the Bangla Version of the COVID-19 Anxiety Scale

関谷徳泰さんのブログ記事(測定不変性(measurement invariance)、測定等価性(measurement equivalence) | Dr.Clover's Computer Clinic)では次のように例が挙げられていた。

統計量をどこまで表記するかは場合に拠りけりのようだが、CFIを優先すべきであるようだ。

![共分散構造分析[R編] 共分散構造分析[R編]](https://m.media-amazon.com/images/I/51PmU3iuLyL.jpg)