デルタ法による間接効果の推定値はサンプルサイズが小さい場合には、正規分布を仮定したz値を使った検定には不向きとなる。その一つの解決策として、モンテカルロ検定に基づく間接効果の推定と信頼区間の推定がある。利用するのはsemToolsパッケージである。
この方法が提案された論文はこちら。
分析に使う例
lavaanのチュートリアルにある例題を利用する。以前のエントリでも利用しているので、内容についてこちらを参照のこと。
library(lavaan)
set.seed(1234)
X <- rnorm(100)
M <- 0.5*X + rnorm(100)
Y <- 0.7*M + rnorm(100)
Data <- data.frame(X = X, Y = Y, M = M)
model <- ' # direct effect
Y ~ c*X
# mediator
M ~ a*X
Y ~ b*M
# indirect effect (a*b)
ab := a*b
# total effect
total := c + (a*b) '
fit <- sem(model, data = Data)
モンテカルロ検定に基づく間接効果の推定
基本的にはリンク先と同じように書いていけばよい。 https://www.rdocumentation.org/packages/semTools/versions/0.3-0/topics/monteCarloMed
# 間接効果の表現を書く med <- 'a*b' #解析結果のパラメータ値 aparam <- coef(fit)[["a"]] bparam <- coef(fit)[["b"]] #分析からの漸近的共分散行列(Asymptotic Covariance Matrix) AC <- vcov(fit)[c(2,3),c(2,3)]
ここの行列に入れるのはaとbだけ入れればよい。vcov(fit)で生成される共分散行列はどうなっているかというと、以下のようになっている。
c a b Y~~Y M~~M c 0.011 a 0.000 0.011 b -0.004 0.000 0.009 Y~~Y 0.000 0.000 0.000 0.016 M~~M 0.000 0.000 0.000 0.000 0.022
この中で必要なのは2行目、3行目と2列目、3列目の行列である。
ということで、それだけ取り出すのは[c(2,3),c(2,3)]と書き加えればよい。Rの行列操作で行う方法の一つである。下部に一番基本形を書いておく。
同じように[["a"]]はaだけ取り出すという意味である。
計算とプロット
monteCarloMed(med, coef1=aparam, coef2=bparam,
outputValues=FALSE, ACM=AC, plot=TRUE)
outputValuesはFALSEにする。TRUEにすると反復計算の結果がすべて表示されるため大変なことになる。
plotはTRUEに。そのプロットを下記に表示する。
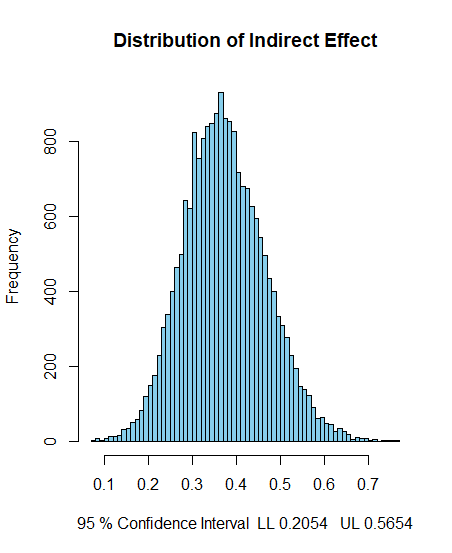
使用したサンプルデータが正規分布の乱数を利用したものだけあって、割ときれいな正規分布のプロットになっている。実際のデータであれば、95%信頼区間の左右の広がりが違っていたりする。
結果は以下のように表示される。
$`Point Estimate`
[1] 0.3735964
$`95% Confidence Interval`
LL 0.2054
UL 0.5654
正規分布の乱数で作ったデータで比較してもあまりありがたみがないが、デルタ法では推定値は0.374[95%CI: 0.193 - 0.554]であった。ほとんど同じ値となっている。
行列から任意の行と列を抜き出す
> mat <- matrix(1:9, nrow=3, dimnames=list(c('r1', 'r2', 'r3'), c('c1', 'c2', 'c3')))
> mat
c1 c2 c3
r1 1 4 7
r2 2 5 8
r3 3 6 9
> mat[c(2, 3), c(2, 3)]
c2 c3
r2 5 8
r3 6 9