https://www.math.mcgill.ca/yyang/comp/notes/note4code.R
library(glmnet)
このパッケージで使用されるデフォルトのモデルは、Guassian線形モデルまたは最小二乗法である。説明のためにあらかじめ作成したデータセットをロードする。ユーザは自分のデータをロードすることも、ワークスペースに保存されているデータを使用することもできる。
pacman::p_install_gh("emeryyi/gglasso")
library(gglasso) data(bardet) x<- bardet[["x"]] y<- bardet[["y"]]
glmnetの最も基本的な呼び出しを使ってモデルをフィットさせる。
fit = glmnet(x, y)
"fit "はglmnetクラスのオブジェクトで、フィットされたモデルの関連情報をすべて含んでいる。我々は、ユーザが直接成分を抽出することを推奨していない。その代わりに、plot、print、coef、predictなどの様々なメソッドがオブジェクトに提供され、これらのタスクをよりエレガントに実行することができる。
plot関数を実行すれば、係数を可視化することができる:
plot(fit)

各曲線は変数に対応する。これは、λが変化する時の係数ベクトル全体の「1-norm」に対する係数のパスを示している。上の軸は、現在のλにおける非ゼロ係数の数を示しており、これはlassoの有効自由度(df)となる。ユーザーは、曲線に注釈を付けることもでき、これはplotコマンドでlabel = TRUEを設定することで可能だ。
オブジェクト名を入力するか、print機能を使えば、各ステップでのglmnetパスの概要が表示される:
print(fit)
Call: glmnet(x = x, y = y)
Df %Dev Lambda
1 0 0.00 0.097200
2 1 7.73 0.088560
3 1 14.16 0.080690
4 1 19.49 0.073520
5 1 23.91 0.066990
6 1 27.59 0.061040
7 2 31.42 0.055620
8 2 36.05 0.050680
9 4 40.59 0.046180
10 6 45.05 0.042070
...
左からゼロでない係数の数(Df)、-log(尤度)の値(%dev)、ラムダの値(Lambda)が表示される。デフォルトではglmnetは100個のλの値を要求するが、%dev%がλの値から次のλの値まで十分に変化しない場合、プログラムは早期に停止する(典型的にはパスの終わり近く)。
配列の範囲内の1つ以上のラムダで実際の係数を得ることができる:
coef0 = coef(fit,s=0.1)
関数glmnetは、ユーザーが選択できる一連のモデルを返す。多くの場合、ユーザーはソフトウェアがその中から1つを選択することを好むかもしれない。クロスバリデーションは、おそらくそのタスクのための最もシンプルで最も広く使われている方法である。
cv.glmnetは、プロットや予測などの様々なサポートメソッドとともに、クロスバリデーションを行うためのメイン関数です。ここでは、前に読み込んだサンプルデータを使っている。
cvfit = cv.glmnet(x, y)
cv.glmnetは、cv.glmnetオブジェクトを返しますが、これはここでは "cvfit"であり、クロスバリデーション適合のすべての成分を含むリストです。glmnetに関しては、選択されたλの値を見る以外には、ユーザが直接成分を抽出することは推奨しません。このパッケージは、潜在的なタスクのためによく設計された関数を提供する。
オブジェクトをプロットできる。
plot(cvfit)

クロスバリデーション曲線(赤い点線)、ラムダ配列に沿った上下の標準偏差曲線(エラーバー)が含まれる。選択された2つのラムダは縦の点線で示されている(下図参照)。
選択されたラムダと対応する係数を見ることができる。例えば、
cvfit$lambda.min
[1] 0.001226463
lambda.minは、クロスバリデーションの平均誤差を最小にするλの値である。もう1つの保存されたλはλ.1seで、これは誤差が最小の標準誤差の1つ以内に収まるような最も正則化されたモデルを与える。これを使うには、上記のlambda.minをlambda.1seに置き換えるだけでよい。
coef1 = coef(cvfit, s = "lambda.min")
係数はスパース行列形式で表現されていることに注意。その理由は、正則化パスに沿った解はスパースであることが多いため、スパースフォーマットを使用する方が時間的にも空間的にも効率的だからだ。スパースでない形式を好む場合は、出力をas.matrix()にパイプを通して出力する。
予測は、フィットされたcv.glmnetオブジェクトに基づいて行うことができる。おもちゃの例を見てみよう。
predict(cvfit, newx = x[1:5,], s = "lambda.min")
lambda.min
[1,] 8.406305 [2,] 8.353781 [3,] 8.377383 [4,] 8.294958 [5,] 8.379072
newxは新しい入力行列を表し、sは前回と同様、予測が行われるλの値(複数可)である。
adaptive lasso
ペナルティ・ファクターの例
[penalty.factor]引数により、各係数に別々のペナルティ係数を適用することができる。デフォルトは各パラメータに対して1だが、他の値を指定することもできる。特に、[penalty.factor]が0に等しい変数は、全くペナルティを受けません!v_jがj番目の変数の制約を表すとする。
ペナルティ制約は、内部的にnvarsの合計になるように再スケーリングされることに注意。
これは、変数に対する予備知識や好みがある場合に非常に便利だ。多くの場合、ある変数が非常に重要で、それを常に保持したい場合があり、これは対応する制約係数を0に設定することで実現できる: v_j は、ペナルティ係数を0に設定することで、ペナルティ係数を0にすることができる:
p.fac = rep(1, 120) p.fac[c(31, 74, 111)] = 0 pfit = glmnet(x, y, penalty.factor = p.fac) plot(pfit, xvar="lambda", label = TRUE)
その他の便利な引数をいくつか挙げる: [exclude]は、特定の変数をモデルにしないようにすることができる。もちろん、単純にxからこれらの変数を除外することもできるが、除外された変数をゼロに設定するだけで、係数の完全なベクトルを返すので、excludeの方が便利な場合もある。FALSEの場合、切片は強制的にゼロになる。
Adaptive lassoの例
adaptive lassoは一貫性のある第1段階を必要とする。 Zou (2006)はOLSかridge第一段lassoを推奨している。
thelasso.cv<-cv.glmnet(x,y,family = "gaussian",alpha=1)
第1段階の係数から第2段階の重みを求める coef() はスパース行列
bhat<-as.matrix(coef(thelasso.cv,s="lambda.1se"))[-1,1]
if(all(bhat==0)){
bhat<-rep(.Machine$double.eps*2,length(bhat))
}
もしbhatがすべてゼロなら、すべてにゼロに極めて近いウェイトを割り当てる。これは、第2ステージのすべてにゼロのペナルティを課すことに等しい。
adaptive lasso weight
adpen<-(1/pmax(abs(bhat),.Machine$double.eps))
第2段階のlasso(adaptive lasso)
m_adlasso <- glmnet(x,y,family = "gaussian",alpha=1,exclude=which(bhat==0), penalty.factor=adpen) plot(m_adlasso)

エラスティックネットelastic-net
エラスティック・ネットの例
glmnetには、ユーザーがフィットをカスタマイズするための様々なオプションが用意されている。ここではよく使われるオプションをいくつか紹介するが、これらはglmnet関数で指定することができる。
[family="gaussian"]はglmnet関数のデフォルトのfamilyオプションだ。
[alpha]は弾性ネットの混合パラメータalphaで、alphaの範囲は[0,1]です。alpha=1はlasso(デフォルト)、alpha=0はリッジになる。
[nlambda] はシーケンス中のラムダ値の数であり、デフォルトは100ですが、[0,1]の範囲もある。
例として、α=0.2(よりリッジ回帰に近い)を設定し、オブザベーションの後半に2倍の重みを与える。 ここでの表示が長くなりすぎるのを避けるために、nlambdaを20に設定する。しかし実際には、λの値の数は100(デフォルト)以上にすることが推奨される。ほとんどの場合、アルゴリズムで使用されるウォーム・スタート warm-starts のために、余分なコストがかからず、非線形モデルではよりよい収束特性につながる。
fit = glmnet(x, y, alpha = 0.2, family="gaussian") plot(fit, xvar = "lambda", label = TRUE)
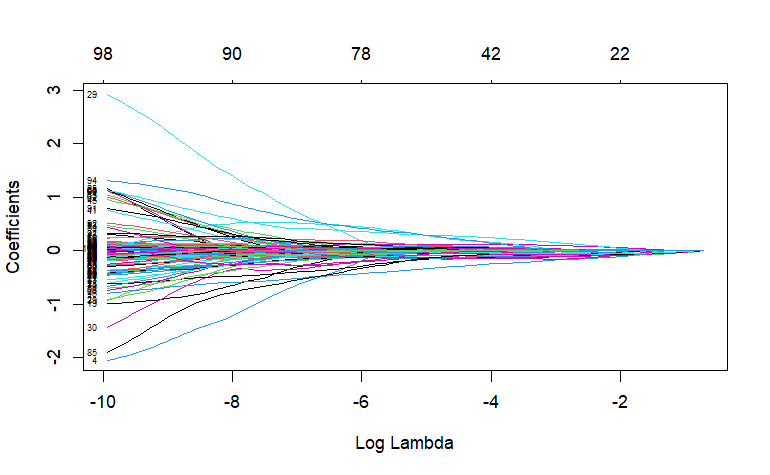
グループlasso
グループlassoの例
install.packages("gglasso", repos = "http://cran.us.r-project.org")
https://cran.r-project.org/web/packages/gglasso/index.html
group-lasso ペナルティ付き最小二乗法、ロジスティック回帰、Huberized SVM、二乗SVMの解経路を効率的に計算するための統一アルゴリズム、blockwise-majorization-descent (BMD)。 このパッケージは、Yang, Y. and Zou, H. (2015) DOI: <doi:10.1007/s11222-014-9498-5> の実装だ。
library(gglasso)
bardetデータセットの読み込み
data(bardet)
20のグループを定義する
group1 <- rep(1:20,each=5)
ペナルティ付き最小二乗"group lasso"にフィットさせる。
m1 <- gglasso(x=bardet$x,y=bardet$y,group=group1,loss="ls")
plot(m1)
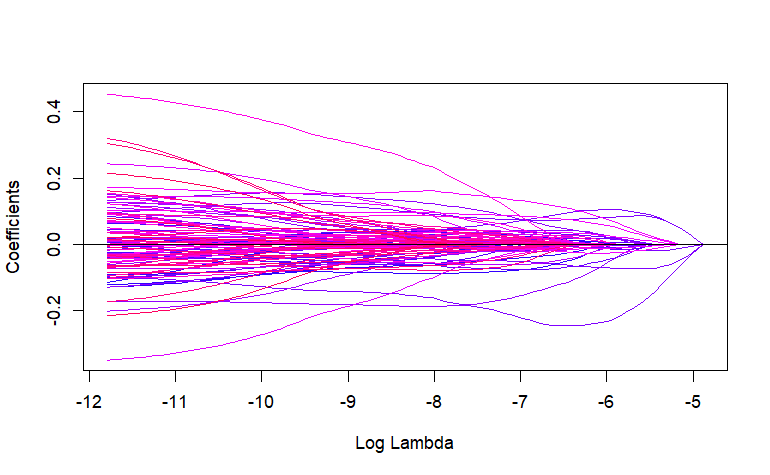
ペナルティ付きグループLassoでls回帰を用いた5-fold クロスバリデーション
cv <- cv.gglasso(x=bardet$x, y=bardet$y, group=group1, loss="ls", pred.loss="L2", lambda.factor=0.05, nfolds=5)
plot(cv)

colon データセットを読み込む
data(colon)
グループ指標を定義する
group2 <- rep(1:20,each=5)
グループlassoペナルティ付きロジスティック回帰にフィット
m2 <- gglasso(x=colon$x,y=colon$y,group=group2,loss="logit")
plot(m2)
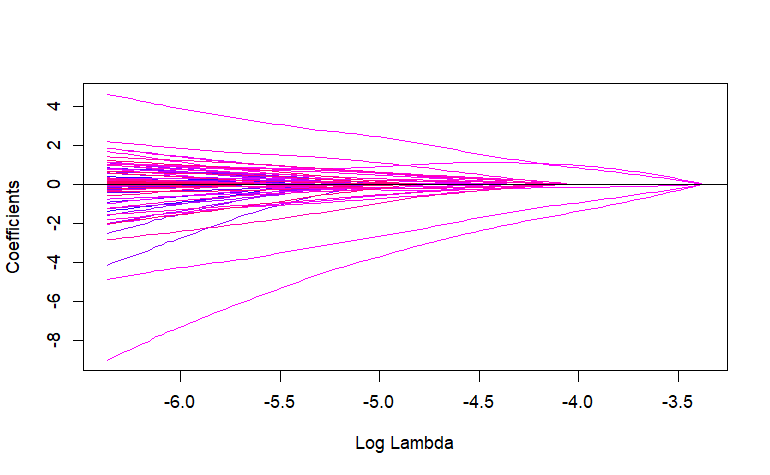
グループlassoペナルティ付きロジスティック回帰を用いた5-foldクロスバリデーション
cv2 <- cv.gglasso(x=colon$x, y=colon$y, group=group2, loss="logit", pred.loss="misclass", lambda.factor=0.05, nfolds=5)
plot(cv2)

λ=λ.1seにおける係数
pre = coef(cv$gglasso.fit, s = cv$lambda.1se)
シミュレーション・デモ-1
ロジスティック回帰と最小二乗法におけるlasso、adaptive lasso、scad、mcpの比較
Rライブラリをロード
glmnet: LASSO、adaptive LASSO、elastic net ペナルティ付き最小2乗法およびロジスティック回帰 (すべてポアソン、多項式、コックスモデルをサポート)
ncvreg: SCAD および MCP ペナルティ化最小2乗法およびロジスティック回帰用
library(glmnet) library(ncvreg) library(MASS)
PART I 最小二乗法
n=100 p=200
真のベータ
truebeta <- c(4,4,4,-6*sqrt(2),4/3,rep(0,p-5))
誤差分散
sigma2 <- 0.3
XjとXkの間の共分散はcov(X_j, X_k) = rho^|i-j|共分散行列である。
covmat <- function(rho, p) {
rho^(abs(outer(seq(p), seq(p), "-")))
}
rho = 0.1、Xの共分散行列を生成する。
sigma <- covmat(0.1,p)
X ~ N(0, sigma)
epsilon ~ N(0, sigma2)
真のモデル: y = x*truebeta + epsilon
x <- mvrnorm(n,rep(0,p),sigma) epsilon <- rnorm(n,0,sd=sqrt(sigma2)) y <- x %*% truebeta + epsilon
lassoをフィットさせ、5-fold CVでラムダを選択する。
cvfit <- cv.glmnet(x = x, y = y, alpha = 1, family = "gaussian") plot(cvfit)

CVで選ばれたラムダ
cvfit$lambda.min
[1] 0.0821079
解決パスをプロットする
plot(cvfit$glmnet.fit,label=TRUE,xvar="lambda")

λ.minのプロット
abline(v=log(cvfit$lambda.min),lty=1)
λ.1seをプロットする
abline(v=log(cvfit$lambda.1se),lty=2)
model_compare <- matrix(NA, nrow=5, ncol=p,
dimnames=list(c("true model",
"lasso","adaptive lasso","mcp","scad"),
paste("V",seq(p),sep="")))
真のモデルを保存する
model_compare[1, ] <- truebeta
lassoによって推定された係数
cvfit <- cv.glmnet(x = x, y = y, alpha = 1, family = "gaussian") tmp <- cvfit$glmnet.fit$beta lasso_beta <- as.matrix(tmp[,cvfit$lambda==cvfit$lambda.min]) model_compare[2, ] <- lasso_beta
重みを計算し、adaptive lassoにフィットさせる
weight = 1/(lasso_beta)^2
推定係数がゼロの場合、対応するウェイトがInfとなり、数値誤差を防ぐため、Infを大きな数値(例えば1e6)に変換する。
weight[weight==Inf] = 1e6
cvfit <- cv.glmnet(x = x, y = y, alpha = 1,
family = "gaussian",
nfolds = 5, penalty.factor=weight)
Adaptive Lassoによって推定された係数
tmp <- cvfit$glmnet.fit$beta adaptive_lasso_beta <- as.matrix(tmp[,cvfit$lambda==cvfit$lambda.min]) model_compare[3, ] <- adaptive_lasso_beta
mcpによって推定された係数
cvfit <- cv.ncvreg(X = x, y = y, penalty = "MCP", family = "gaussian") mcp_beta <- cvfit$fit$beta[, cvfit$min] model_compare[4, ] <- mcp_beta[-1]
scadによって推定された係数
cvfit <- cv.ncvreg(X = x, y = y, penalty = "SCAD", family = "gaussian") scad_beta <- cvfit$fit$beta[, cvfit$min] model_compare[5, ] <- scad_beta[-1]
4つの方法で推定された係数を比較すると、lasso over-selected、adaptive lasso、scad、mcpが問題を解決していることがわかる。
model_compare
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
true model 4 4.000000000 4 -8.485281 1.333333 0 0 0 0 0
lasso 0 -0.022023796 0 0.000000 0.000000 0 0 0 0 0
adaptive lasso 0 0.000000000 0 0.000000 0.000000 0 0 0 0 0
mcp 0 0.000000000 0 0.000000 0.000000 0 0 0 0 0
scad 0 -0.004545409 0 0.000000 0.000000 0 0 0 0 0
V11 V12 V13 V14 V15 V16
true model 0.000000000 0.00000000 0.0000000 0.0000000 0.000000 0
lasso 0.006311609 0.00000000 0.0000000 0.0000000 0.000000 0
adaptive lasso 0.000000000 0.00000000 0.0000000 0.0000000 0.000000 0
mcp 0.000000000 -0.08207367 -0.1647326 0.2088095 -1.000567 0
scad 0.000000000 0.00000000 0.0000000 0.0000000 -1.033549 0
V17 V18 V19 V20 V21 V22 V23
true model 0.00000000 0.00000000 0 0 0 0.0000000000 0.00000000
lasso 0.00000000 -0.01762063 0 0 0 0.0001952964 -0.05328850
adaptive lasso 0.00000000 0.00000000 0 0 0 0.0000000000 0.00000000
mcp 0.03504008 0.00000000 0 0 0 0.0000000000 0.00000000
scad 0.00000000 0.00000000 0 0 0 0.0000000000 -0.08488739
V24 V25 V26 V27 V28 V29 V30
true model 0.0000000 0.0000000 0.00000000 0 0.0000000 0 0.0000000
lasso 0.0000000 -0.5648782 0.06002556 0 0.0000000 0 -0.3371098
adaptive lasso 0.0000000 -1.0114037 0.00000000 0 0.0000000 0 0.0000000
mcp -0.4379709 0.0000000 0.00000000 0 -0.1994319 0 0.0000000
scad 0.0000000 0.0000000 0.00000000 0 0.0000000 0 0.0000000
V31 V32 V33 V34 V35 V36 V37 V38 V39 V40 V41
true model 0.0000000 0 0.0000000 0 0 0.0000000 0 0 0 0 0
lasso -0.1165544 0 0.0000000 0 0 0.0482393 0 0 0 0 0
adaptive lasso 0.0000000 0 0.0000000 0 0 0.0000000 0 0 0 0 0
mcp -0.4561359 0 -0.1259705 0 0 0.1201394 0 0 0 0 0
scad 0.0000000 0 0.0000000 0 0 0.0000000 0 0 0 0 0
V42 V43 V44 V45 V46 V47 V48 V49 V50
true model 0 0 0.000000000 0 0.0000000 0.0000000 0 0 0.0000000
lasso 0 0 0.009982214 0 -0.2897772 0.0000000 0 0 0.0000000
adaptive lasso 0 0 0.000000000 0 -0.6616183 0.0000000 0 0 0.0000000
mcp 0 0 0.000000000 0 -0.4648836 0.1156758 0 0 -0.1438189
scad 0 0 0.000000000 0 -0.1163973 0.0000000 0 0 0.0000000
V51 V52 V53 V54 V55 V56 V57 V58 V59
true model 0.0000000 0 0.00000000 0.00000000 0 0 0 0.00000000 0
lasso 0.0000000 0 0.05634673 0.13775797 0 0 0 0.00000000 0
adaptive lasso 0.0000000 0 0.00000000 0.09460430 0 0 0 0.00000000 0
mcp 0.0000000 0 0.07499241 0.11837706 0 0 0 -0.05640483 0
scad -0.1008567 0 0.00000000 0.08972644 0 0 0 0.00000000 0
V60 V61 V62 V63 V64 V65 V66 V67 V68 V69 V70
true model 0 0 0 0.00000000 0 0.0000000 0.00000000 0 0 0 0
lasso 0 0 0 0.00000000 0 0.2563789 -0.12041928 0 0 0 0
adaptive lasso 0 0 0 0.00000000 0 0.2957797 0.00000000 0 0 0 0
mcp 0 0 0 0.02528918 0 0.3411462 0.00000000 0 0 0 0
scad 0 0 0 0.00000000 0 0.1248035 -0.03840615 0 0 0 0
V71 V72 V73 V74 V75 V76 V77 V78 V79
true model 0 0 0.00000000 0 0 0.00000000 0.0000000 0.00000000 0
lasso 0 0 0.05161909 0 0 0.00000000 0.0000000 0.04393271 0
adaptive lasso 0 0 0.00000000 0 0 0.00000000 0.0000000 0.00000000 0
mcp 0 0 0.07055282 0 0 0.00000000 0.0652188 0.12140394 0
scad 0 0 0.00000000 0 0 -0.06290252 0.0000000 0.00000000 0
V80 V81 V82 V83 V84 V85 V86 V87 V88 V89
true model 0 0 0 0 0 0 0.00000000 0.000000000 0 0.0000000
lasso 0 0 0 0 0 0 0.00000000 -0.001347216 0 0.2044193
adaptive lasso 0 0 0 0 0 0 0.00000000 0.000000000 0 0.1554728
mcp 0 0 0 0 0 0 0.04738824 0.000000000 0 0.4817615
scad 0 0 0 0 0 0 0.00000000 0.000000000 0 0.0000000
V90 V91 V92 V93 V94 V95 V96 V97 V98 V99 V100 V101
true model 0 0 0 0 0.00000000 0 0 0 0 0 0.00000000 0
lasso 0 0 0 0 0.07206646 0 0 0 0 0 0.06736159 0
adaptive lasso 0 0 0 0 0.00000000 0 0 0 0 0 0.00000000 0
mcp 0 0 0 0 0.44369829 0 0 0 0 0 0.14673570 0
scad 0 0 0 0 0.00000000 0 0 0 0 0 0.00000000 0
V102 V103 V104 V105 V106 V107 V108 V109 V110 V111
true model 0.000000000 0 0 0 0 0 0 0 0 0.000000000
lasso -0.022023796 0 0 0 0 0 0 0 0 0.006311609
adaptive lasso 0.000000000 0 0 0 0 0 0 0 0 0.000000000
mcp 0.000000000 0 0 0 0 0 0 0 0 0.000000000
scad -0.004545409 0 0 0 0 0 0 0 0 0.000000000
V112 V113 V114 V115 V116 V117
true model 0.00000000 0.0000000 0.0000000 0.000000 0 0.00000000
lasso 0.00000000 0.0000000 0.0000000 0.000000 0 0.00000000
adaptive lasso 0.00000000 0.0000000 0.0000000 0.000000 0 0.00000000
mcp -0.08207367 -0.1647326 0.2088095 -1.000567 0 0.03504008
scad 0.00000000 0.0000000 0.0000000 -1.033549 0 0.00000000
V118 V119 V120 V121 V122 V123 V124
true model 0.00000000 0 0 0 0.0000000000 0.00000000 0.0000000
lasso -0.01762063 0 0 0 0.0001952964 -0.05328850 0.0000000
adaptive lasso 0.00000000 0 0 0 0.0000000000 0.00000000 0.0000000
mcp 0.00000000 0 0 0 0.0000000000 0.00000000 -0.4379709
scad 0.00000000 0 0 0 0.0000000000 -0.08488739 0.0000000
V125 V126 V127 V128 V129 V130 V131
true model 0.0000000 0.00000000 0 0.0000000 0 0.0000000 0.0000000
lasso -0.5648782 0.06002556 0 0.0000000 0 -0.3371098 -0.1165544
adaptive lasso -1.0114037 0.00000000 0 0.0000000 0 0.0000000 0.0000000
mcp 0.0000000 0.00000000 0 -0.1994319 0 0.0000000 -0.4561359
scad 0.0000000 0.00000000 0 0.0000000 0 0.0000000 0.0000000
V132 V133 V134 V135 V136 V137 V138 V139 V140 V141
true model 0 0.0000000 0 0 0.0000000 0 0 0 0 0
lasso 0 0.0000000 0 0 0.0482393 0 0 0 0 0
adaptive lasso 0 0.0000000 0 0 0.0000000 0 0 0 0 0
mcp 0 -0.1259705 0 0 0.1201394 0 0 0 0 0
scad 0 0.0000000 0 0 0.0000000 0 0 0 0 0
V142 V143 V144 V145 V146 V147 V148 V149
true model 0 0 0.000000000 0 0.0000000 0.0000000 0 0
lasso 0 0 0.009982214 0 -0.2897772 0.0000000 0 0
adaptive lasso 0 0 0.000000000 0 -0.6616183 0.0000000 0 0
mcp 0 0 0.000000000 0 -0.4648836 0.1156758 0 0
scad 0 0 0.000000000 0 -0.1163973 0.0000000 0 0
V150 V151 V152 V153 V154 V155 V156 V157
true model 0.0000000 0.0000000 0 0.00000000 0.00000000 0 0 0
lasso 0.0000000 0.0000000 0 0.05634673 0.13775797 0 0 0
adaptive lasso 0.0000000 0.0000000 0 0.00000000 0.09460430 0 0 0
mcp -0.1438189 0.0000000 0 0.07499241 0.11837706 0 0 0
scad 0.0000000 -0.1008567 0 0.00000000 0.08972644 0 0 0
V158 V159 V160 V161 V162 V163 V164 V165
true model 0.00000000 0 0 0 0 0.00000000 0 0.0000000
lasso 0.00000000 0 0 0 0 0.00000000 0 0.2563789
adaptive lasso 0.00000000 0 0 0 0 0.00000000 0 0.2957797
mcp -0.05640483 0 0 0 0 0.02528918 0 0.3411462
scad 0.00000000 0 0 0 0 0.00000000 0 0.1248035
V166 V167 V168 V169 V170 V171 V172 V173 V174 V175
true model 0.00000000 0 0 0 0 0 0 0.00000000 0 0
lasso -0.12041928 0 0 0 0 0 0 0.05161909 0 0
adaptive lasso 0.00000000 0 0 0 0 0 0 0.00000000 0 0
mcp 0.00000000 0 0 0 0 0 0 0.07055282 0 0
scad -0.03840615 0 0 0 0 0 0 0.00000000 0 0
V176 V177 V178 V179 V180 V181 V182 V183 V184
true model 0.00000000 0.0000000 0.00000000 0 0 0 0 0 0
lasso 0.00000000 0.0000000 0.04393271 0 0 0 0 0 0
adaptive lasso 0.00000000 0.0000000 0.00000000 0 0 0 0 0 0
mcp 0.00000000 0.0652188 0.12140394 0 0 0 0 0 0
scad -0.06290252 0.0000000 0.00000000 0 0 0 0 0 0
V185 V186 V187 V188 V189 V190 V191 V192 V193
true model 0 0.00000000 0.000000000 0 0.0000000 0 0 0 0
lasso 0 0.00000000 -0.001347216 0 0.2044193 0 0 0 0
adaptive lasso 0 0.00000000 0.000000000 0 0.1554728 0 0 0 0
mcp 0 0.04738824 0.000000000 0 0.4817615 0 0 0 0
scad 0 0.00000000 0.000000000 0 0.0000000 0 0 0 0
V194 V195 V196 V197 V198 V199 V200
true model 0.00000000 0 0 0 0 0 0.00000000
lasso 0.07206646 0 0 0 0 0 0.06736159
adaptive lasso 0.00000000 0 0 0 0 0 0.00000000
mcp 0.44369829 0 0 0 0 0 0.14673570
scad 0.00000000 0 0 0 0 0 0.00000000
シミュレーション・デモ-2
PART II ロジスティック回帰
データを生成する
n <- 200 p <- 8
真のベータ
truebeta <- c(6,3.5,0,5,rep(0,p-4)) truebeta
真のモデルからxとyを生成 真のモデル: P(y=1|x) = exp(xtruebeta)/(exp(xtruebeta)+1)
x <- matrix(rnorm(n*p), n, p)
feta <- x %*% truebeta
fprob <- ifelse(feta < 0, exp(feta)/(1+exp(feta)), 1/(1 + exp(-feta)))
y <- rbinom(n, 1, fprob)
model_compare <- matrix(NA, nrow=5, ncol=p,
dimnames=list(c("true model","lasso",
"adaptive lasso","mcp","scad"),
paste("V",seq(p),sep="")))
真のモデルを保存する
model_compare[1, ] <- truebeta
lassoのケース
cv.glmfit ラッソモデルをフィットし、ラムダの選択にクロス検証を使用する。
family = "binomial", ロジスティック回帰
family = "gaussian", 最小二乗法
alphaはL1ペナルティ項の度合いを制御する、
alpha = 1, lasso、
alpha = 0, ridge、
alpha = (0,1), 弾性ネット
nfolds = 5, 5回クロスバリデーション(CV)
cvfit <- cv.glmnet(x = x, y = y, alpha = 1, family = "binomial", nfolds = 5)
CV結果をプロットする
左の縦線(λ.min)は、最小の偏差を与えるλに対応する。
右の縦線(λ.1se)は、1標準偏差ルールのλに対応する。
plot(cvfit)

解決パスをプロットする
plot(cvfit$glmnet.fit,label=TRUE,xvar="lambda")

plot(cvfit$glmnet.fit,label=TRUE,xvar="lambda")

λ.minをプロットする
abline(v=log(cvfit$lambda.min),lty=1)
λ.1seをプロットする
abline(v=log(cvfit$lambda.1se),lty=2)
各列はラムダ値の推定値を表す。
tmp <- cvfit$glmnet.fit$beta tmp
CVが選択したλ.minに対応するベータ値
lasso_beta <- as.matrix(tmp[,cvfit$lambda==cvfit$lambda.min]) model_compare[2, ] <- lasso_beta
重みを計算し、Adaptive Lassoをフィットさせる
weight = 1/(lasso_beta)^2
推定係数がゼロの場合、対応するウェイトがInfとなり、数値誤差を防ぐため、Infを大きな数値(例えば1e6)に変換する。
weight[weight==Inf] = 1e6
cvfit <- cv.glmnet(x = x, y = y, alpha = 1, family = "binomial",
nfolds = 5, penalty.factor=weight)
Adaptive Lassoによって推定された係数
tmp <- cvfit$glmnet.fit$beta adaptive_lasso_beta <- as.matrix(tmp[,cvfit$lambda==cvfit$lambda.min]) model_compare[3, ] <- adaptive_lasso_beta
mcpによって推定された係数
cvfit <- cv.ncvreg(X = x, y = y, penalty = "MCP", family = "binomial") mcp_beta <- cvfit$fit$beta[, cvfit$min] model_compare[4, ] <- mcp_beta[-1]
scadによって推定された係数
cvfit <- cv.ncvreg(X = x, y = y, penalty = "SCAD", family = "binomial") scad_beta <- cvfit$fit$beta[, cvfit$min] model_compare[5, ] <- scad_beta[-1]
4つの方法から推定された係数を比較する。
model_compare
V1 V2 V3 V4 V5 V6 V7
true model NA NA NA NA NA NA NA
lasso 4.467528 2.375300 0 3.096532 0 -0.07315415 -0.3499353
adaptive lasso 4.664295 2.480856 0 3.335227 0 0.00000000 0.0000000
mcp 4.459921 2.533341 0 3.366028 0 0.00000000 0.0000000
scad 4.808539 2.744814 0 3.615375 0 0.00000000 0.0000000
V8
true model NA
lasso 0.3801377
adaptive lasso 0.0000000
mcp 0.0000000
scad 0.0000000