こちらの3章の翻訳。
https://cran.r-project.org/web/packages/PSweight/vignettes/vignette.pdf
3.パッケージの概要
PSweightパッケージには、観察研究のデザインと分析に特化した2つのモジュールが含まれている。 デザインモジュールは、傾向スコアモデルと重み付けされた対象集団の妥当性を、結果データを使用する前に評価するための診断機能を提供する。解析モジュールは、セクション2で議論した因果関係の推定値を推定する機能を提供する。この2つのモジュールについて、以下に簡単に説明する。
3.1. Design モジュール PSweightはSumStat()関数を用いて、推定された傾向スコアの分布を可視化し、異なる重み付けスキームにおける共変量のバランスを評価し、重み付けされた対象集団の特徴を示すことができます。以下のコードスニペットを使用する。
SumStat(ps.formula, ps.estimate = NULL, trtgrp = NULL, Z = NULL, covM = NULL, + zname = NULL, xname = NULL, data = NULL, weight = "overlap", delta = 0, + method = "glm", ps.control = list())
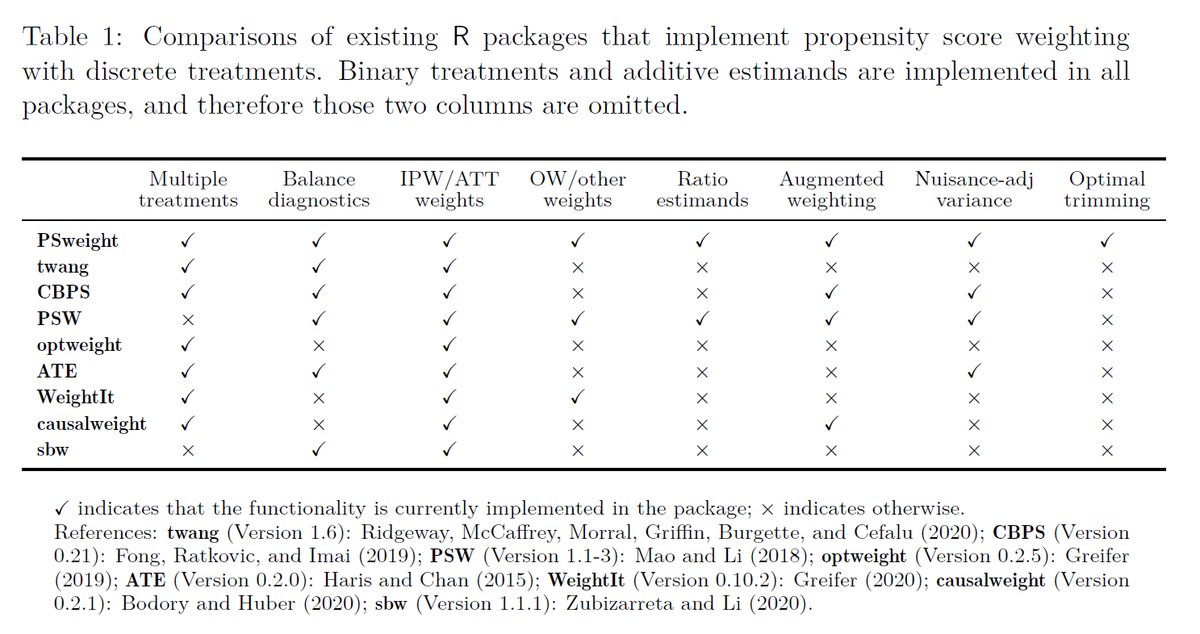
デフォルトでは、引数ps.formulaを通じて、(一般化)傾向スコアが(多項)ロジスティック回帰によって推定される。あるいは,gbmパッケージのgbm()関数(Greenwell, Boehmke, Cunningham, and Developers 2019)またはSuperLearnerパッケージのSuperLearner()関数(Polly, LeDell, Kennedy, and van der Laan 2019)も method = "gbm" または method = "SuperLearner" で呼ぶことができる。これらの関数の追加のパラメータは、ps.control引数を通じて提供することができる。引数ps.estimateは、外部ルーチンからの推定傾向スコアをサポートする。SumStat()は、推定傾向スコア、各治療群の非加重および加重共変量平均、バランス診断、有効サンプルサイズ(Li and Li (2019)で定義)を持つSumStatオブジェクトを生成する。そして、SumStatオブジェクトを受け取り、治療群別の重み付き共変量平均とASDまたはPSDのいずれかの群間差を要約するsummary.SumStat()関数を提供する。デフォルトのオプションであるweighted.var = TRUEとmetric = "ASD" は、Austin and Stuart (2015) の重み付き標準偏差に基づくASDをもたらす。重み付けされた共変量平均は、トリミングまたはバランシングの重みが適用される対象集団を説明するために、ベースライン特性「表1」を構築するために使用することができる。
summary(object, weighted.var = TRUE, metric = "ASD")
傾向スコアモデルの診断は、plot.SumStat()関数で可視化することができる。これは、SumStatオブジェクトを受け取り、ASDとPSDに基づいたbalance plot (type = "balance")を生成する。垂直破線はthreshold引数で設定でき、default値は0.1に等しくなる。plot.SumStat()関数は、密度プロット(type = "density")も提供することができる。
推定された傾向スコアのヒストグラム(type = "hist")を提供することもできる。しかし,ヒストグラムは、バイナリ処理の場合のみ利用可能である。plot関数は次のように実装されている。
plot(x, type = "balance", weighted.var = TRUE, threshold = 0.1, + metric = "ASD")
設計段階では、傾向スコアのトリミングはPStrim()関数で行うことができる。トリミングの閾値Δはデフォルトで0に設定されている。PStrim()は、可能なすべてのトリミング・ルールの中で、最も統計的に有効な(ペアワイズ)部分集団ATEを与える最適トリミング・ルール (optimal = TRUE)も有効にする。トリミングされたデータセットとトリミングされたケースのサマリーがPStrim()によって返される。 この関数を以下に示す。
PStrim(data, ps.formula = NULL, zname = NULL, ps.estimate = NULL, + delta = 0, optimal = FALSE, method = "glm", ps.control = list())
また、トリミングはSumStat()関数の中でデルタ引数で固定されている。設計モジュールの全関数を表4にまとめた。

- SumStat() 傾向スコアと重み付き共変量バランスの情報を持つSumStatオブジェクトを生成する。
- summary.SumStat() SumStat オブジェクトを要約し、ASD または PSD における治療群別の加重共分散平均と加重または非加重群間差異を返す。
- plot.SumStat() SumStat オブジェクトから傾向スコアまたは重み付き共変量バランスメトリクスの分布をプロットする。
- PStrim() 推定された傾向スコアに基づいてデータセットをトリミングする。
3.2. 解析モジュール
PSweightの解析モジュールには2つの関数がある。PSweight()とsummary.PSweight()である。PSweight() 関数は,対象母集団における平均的な潜在的結果を推定する。
{},および関連する分散共分散行列を推定する。デフォルトでは、経験的なサンドイッチ分散が実装されているが、ブートストラップ分散を得るには、引数 bootstrap = TRUEでブートストラップ分散を得ることができる。重みの引数は "IPW", "treated", "overlap", "matching", "entropy "で、第2節で紹介した重みに相当するものである。PSweight() 関数の各入力引数の詳細な説明は,表5を参照されたい。典型的なPSweight()のコードスニペットは以下の通りである。
PSweight(ps.formula, ps.estimate, trtgrp, zname, yname, data, + weight = "overlap", delta = 0, augmentation = FALSE, bootstrap = FALSE, + R = 50, out.formula = NULL, out.estimate = NULL, family = "gaussian", + ps.method = "glm", ps.control = list(), out.method = "glm", + out.control = list())
designモジュールと同様に、summary.PSweight()関数はPSweightオブジェクトから情報を合成し、統計的推論を行う。典型的なコードスニペットは次のようになる。
summary(object, contrast, type = "DIF", CI = TRUE)
type = "DIF" はデフォルトの引数で,加法的因果関係の対比を指定する。type = "RR" は (2.10) 式のように対数スケールでの対比を指定する。type = "OR" は (2.11) 式のように対数オッズスケールでの対比を指定する。信頼区間とp値は正規近似を用いて求められ、summary.PSweight()関数によって報告される。引数contrastは、contrastベクトルaまたは複数のcontrast行ベクトルからなる行列を表す。contrastが指定されない場合、summary.PSweight()は平均的な潜在的な結果の全てのペアワイズ比較を提供する。デフォルトでは、信頼区間が表示されます (CI = TRUE); 代わりに、CI = FALSEによって、テスト統計とp値を表示することもできる。
