新型コロナ関係の疫学の問題に取り組んでいるブログがあり、読んでいるといろいろと勉強なることが多い。
prevalenceパッケージで似たようなことができるエントリがあった気がしたので、少し調べてみた。
prevalenceパッケージはJAGSとrjagsをインストールする必要がある。インストールは特に難しいところはないが、以前のエントリを参考のこと。
調査から真の有病率をベイズ推定する
神戸市の入院中の患者の抗体検査の疫学調査について分析されている。当の抗体検査は使えた代物ではないようなので、あくまでも例題である。
一般に有病率の推定には二項分布を用いた95%区間の数値が用いられる。この方法にもいくつかやり方があって以前に紹介している(参照)。
検査は100%あたるものではない。感度・特異度が判明していれば、それにらの値を加味して真の有病率を推定することができるのだ。
必要なデータは以下の4つである。
library("prevalence")
Prev <- truePrev(33, 1000, SE = 0.7638, SP = 1, nchains = 4,
burnin = 10000, update = 10000, verbose = FALSE)
Prev
結果。
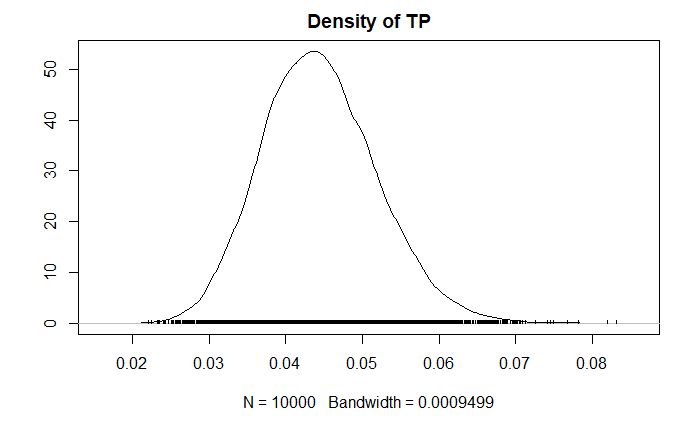
mean median mode sd 2.5% 97.5% TP 0.044 0.044 0.043 0.008 0.031 0.060 SE 0.764 0.764 0.764 0.000 0.764 0.764 SP 1.000 1.000 1.000 0.000 1.000 1.000 BGR statistic = 1.0001 (upper CL = 1.0001) BGR values substantially above 1 indicate lack of convergence
TPがベイズ推定した値である。4.4%(95%CI: 3.1%-6.0%)でガルバッタさんの導いた数値とは違ってはいるが、そういうこともあるのかなと思いつつ、prevalenceパッケージが出している特異度が1.000というところは少し気になる。
オプションとしては、例えば一様分布で感度が0.6-1.0、特異度が0.75-1.0であった場合には下記のように書くようだ。
truePrev(x = 142, n = 742, SE = ~dunif(0.60, 1.00), SP = ~dunif(0.75, 1.00))
プロット
密度プロットとトレースプロットは下記の要領で出力できる。
densplot(Prev, exclude_fixed = TRUE) traceplot(Prev, exclude_fixed = TRUE)
追記(2021/02/12)
補足記事を執筆。